Code
try:
import fysisk_biokemi
print("Already installed")
except ImportError:
%pip install -q "fysisk_biokemi[colab] @ git+https://github.com/au-mbg/fysisk-biokemi.git"Vi har tidligere kigget på et datasæt omkring adsorption spektrummet af chlorophyll, men vi var ikke fokuserede på hvordan vi håndterede dette datasæt.
Det vi nåede frem til var at plotte datasættet, som set herunder
chloro_df = load_dataset("chlorophyll")
bølgelængder = chloro_df['Wavelength(nm)'] # Et array med 501 indgange
adsorption = chloro_df['AdsorptionCoefficient'] # Et array med 501 indgange
fig, ax = plt.subplots()
ax.plot(bølgelængder, adsorption)
ax.set_xlabel('Bølgelængde [nm]')
ax.set_ylabel('Adsorptions koefficient')Text(0, 0.5, 'Adsorptions koefficient')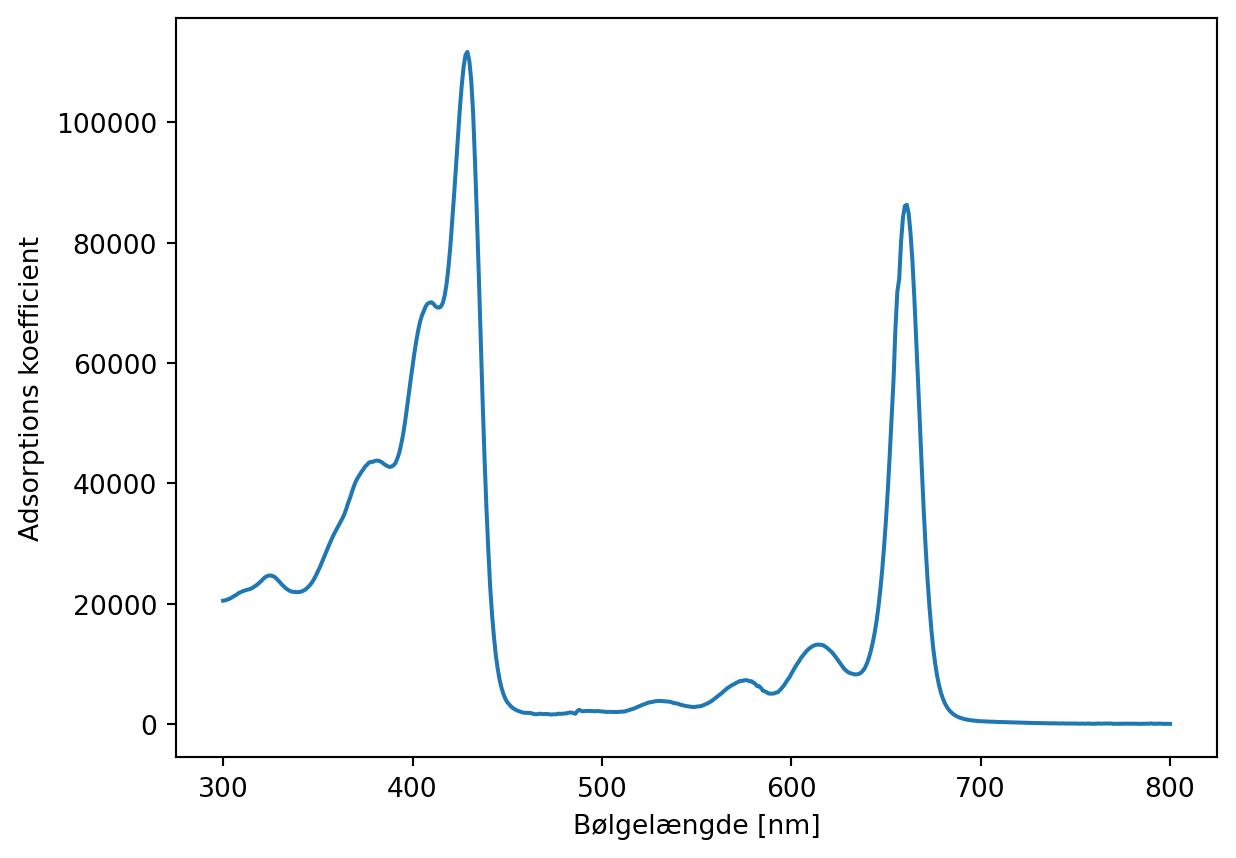
Vi vil denne gang undersøge lidt mere hvordan vi kan arbejde med et datasæt.
Det er vigtigt at vi holder godt styr på vores data, det er ofte smart at bruge en data-type som hjælper os med dette. Vi har tidligere sæt på arrays, som er en mulighed - f.eks.
Men hvis vi har mange størrelser kan det hurtigt blive overskueligt - en anden mulighed er at gemme alle vores størrelser i det samme array
[1 2 3 4]
[ 1 4 9 16]NumPy arrays kan have flere dimensioner, samlet_array har her to dimension (som en matrice). I dette tilfælde beskriver hver række en af vores størrelser. Her er der brugt at : betyder “alle” ved indexing så [0, :] kan læses som første række alle kolonner.
Men det kræver så at vi holde styr hvilken akse der indeholder hvilken størrelse. Nogle gange kan det være fint at holde vores data som np.array men andre gange er det bedre på andre måder.
DataFrameEn sådan anden måde at håndtere et datasæt er ved brug af ny type, nemlig en pandas DataFrame.
Vi kan lave en DataFrame fra vores to arrays sådan her;
Her har vi så tre kolonner, den første er index, den anden er a og den tredje er b.
Chlorophyll datasættet var faktisk også en DataFrame.
Med en DataFrame har vi flere muligheder for indeksering, vi kan f.eks. bruge navnet
Beregn formlen \(2 \times a\) hvor \(a\) kommer fra datasættet ovenfor
Vi kan også lægge to arrays sammen, brug dette til at beregne \(c = a + b\).
Lav et plot af \(a\) mod \(b\) hvor de to størrelser kommer fra DataFrame’en df.
Ofte vil vi ikke skrive vores datasæt direkte ind i kode, men opbevare dem som seperate filer der f.eks. kunne komme fra eksperimentielt udstyr.
Vi kan hente data fra en excel-fil, sådan her
Cellen ovenfor giver en fejl når den bliver kørt fordi den fil vi forsøger at åbne ikke eksistere.
Cellen nedenfor giver stien (path) til et datasæt der er installeret sammen med fysisk_biokemi-pakken.
Brug pandas til at læse filen ind som en dataframe
Udfra det information der er blevet skrevet ud hvad tror du datasættet indeholder? Tænk over følgende
Nu hvor vi har fået hentet datasættet vil vi gerne undersøge det nemmere ved at plotte det.
Færdiggør koden nedenfor
fig, ax = plt.subplots()
ax.plot(df['time'], ..., label='[A]') # Erstat ... med din kode
... # Erstat ... med din kode som plotter tid mod concentration_B
ax.set_ylabel('Concentration') # Sætter navn på y-aksen
ax.set_xlabel('Time') # Sætter navn på x-aksen
ax.legend()# Viser hvad 'label' hver plottet kurve har.Hvad tror du datasættet viser?
Som sagt kan vi også lave beregninger med et datasæt. Udregn \[ [T] = [A] + [B] \] Og færdiggør plottet i cellen nedenfor
concentration_T = ... # Erstat ... med din kode
fig, ax = plt.subplots()
ax.plot(..., ..., label='[T]', color='C2') # Erstat begge ... med din kode.
# Fra før
ax.plot(df['time'], df['concentration_A'], label='[A]')
ax.plot(df['time'], df['concentration_B'], label='[B]')
ax.set_ylabel('Concentration') # Sætter navn på y-aksen
ax.set_xlabel('Time') # Sætter navn på x-aksen
ax.legend() # Viser hvad 'label' hver plottet kurve har.Hvorfor tror du det ser ud som det gør?
---
title: At arbejde med datasæt
categories: ['arrays', 'datasets', 'plotting']
engine: jupyter
format-links:
- text: "Open in Google Colab"
href: "https://colab.research.google.com/github/au-mbg/fysisk-biokemi/blob/built-notebooks/built_notebooks/student/notebook_5.ipynb"
icon: box-arrow-up-right
---
``` {python}
#| eval: false
#| code-fold: true
try:
import fysisk_biokemi
print("Already installed")
except ImportError:
%pip install -q "fysisk_biokemi[colab] @ git+https://github.com/au-mbg/fysisk-biokemi.git"
```
```{python}
import numpy as np
import pandas as pd
from fysisk_biokemi import load_dataset, get_dataset_path
import matplotlib.pyplot as plt
```
Vi har tidligere kigget på et datasæt omkring adsorption spektrummet af chlorophyll,
men vi var ikke fokuserede på hvordan vi håndterede dette datasæt.
Det vi nåede frem til var at plotte datasættet, som set herunder
```{python}
chloro_df = load_dataset("chlorophyll")
bølgelængder = chloro_df['Wavelength(nm)'] # Et array med 501 indgange
adsorption = chloro_df['AdsorptionCoefficient'] # Et array med 501 indgange
fig, ax = plt.subplots()
ax.plot(bølgelængder, adsorption)
ax.set_xlabel('Bølgelængde [nm]')
ax.set_ylabel('Adsorptions koefficient')
```
Vi vil denne gang undersøge lidt mere hvordan vi kan arbejde med et datasæt.
## NumPy arrays som datasæt.
Det er vigtigt at vi holder godt styr på vores data, det er ofte smart at bruge
en data-type som hjælper os med dette. Vi har tidligere sæt på arrays, som er en mulighed - f.eks.
```{python}
a = np.array([1, 2, 3, 4])
b = np.array([1, 4, 9, 16])
```
Men hvis vi har mange størrelser kan det hurtigt blive overskueligt - en anden mulighed
er at gemme alle vores størrelser i det samme array
```{python}
samlet_array = np.array([[1, 2, 3, 4], [1, 4, 9, 16]])
print(samlet_array[0, :]) # Svarer til a
print(samlet_array[1, :]) # Svarer til b
```
::: {.callout-note}
NumPy arrays kan have flere dimensioner, `samlet_array` har her to dimension (som en matrice).
I dette tilfælde beskriver hver række en af vores størrelser. Her er der brugt at `:` betyder "alle"
ved indexing så `[0, :]` kan læses som første række alle kolonner.
:::
Men det kræver så at vi holde styr hvilken akse der indeholder hvilken størrelse.
Nogle gange kan det være fint at holde vores data som `np.array` men andre gange
er det bedre på andre måder.
## Pandas `DataFrame`
En sådan anden måde at håndtere et datasæt er ved brug af ny type, nemlig en
pandas `DataFrame`.
Vi kan lave en `DataFrame` fra vores to arrays sådan her;
```{python}
df = pd.DataFrame({"a": a, "b": b})
print(df)
```
Her har vi så tre kolonner, den første er `index`, den anden er `a` og den tredje er `b`.
::: {.callout-note}
Chlorophyll datasættet var faktisk også en `DataFrame`.
:::
Med en `DataFrame` har vi flere muligheder for indeksering, vi kan f.eks. bruge navnet
```{python}
print(df['a'])
```
### Opgave 1
Beregn formlen $2 \times a$ hvor $a$ kommer fra datasættet ovenfor
```{python}
#| exercise: true
#| eval: false
resultat = ... # Erstat ... med dine kode.
print(resultat)
```
```{python}
#| solution: true
resultat = 2 * df['a']
print(resultat)
```
Vi kan også lægge to arrays sammen, brug dette til at beregne $c = a + b$.
```{python}
#| exercise: true
#| eval: false
c = ... # Erstat ... med din kode
print(c)
```
```{python}
#| solution: true
c = df['a'] + df['b']
print(c)
```
### Opgave 2
Lav et plot af $a$ mod $b$ hvor de to størrelser kommer fra `DataFrame`'en `df`.
```{python}
#| exercise: true
#| eval: false
fig, ax = plt.subplots()
... # Skriv din kode for at plotte.
```
```{python}
#| solution: true
fig, ax = plt.subplots()
ax.plot(df['a'], df['b'])
```
## Datasæt fra filer
Ofte vil vi ikke skrive vores datasæt direkte ind i kode, men opbevare dem som seperate filer
der f.eks. kunne komme fra eksperimentielt udstyr.
Vi kan hente data fra en excel-fil, sådan her
```{python}
#| eval: false
dataset_path = "/sti/til/den/fil/der/har/vores/data.xlsx"
df = pd.read_excel(dataset_path)
```
::: {.callout-warning}
Cellen ovenfor giver en fejl når den bliver kørt fordi den fil vi forsøger at åbne ikke
eksistere.
:::
### Opgave 3: Hent datasæt
Cellen nedenfor giver stien (`path`) til et datasæt der er installeret sammen med `fysisk_biokemi`-pakken.
```{python}
path = get_dataset_path('reversible_reaction')
```
```{python}
#| eval: false
print(path)
```
Brug `pandas` til at læse filen ind som en dataframe
```{python}
#| exercise: true
#| eval: false
df = ... # Erstat ... med din kode.
print(df)
```
```{python}
#| solution: true
df = pd.read_excel(path)
print(df)
```
Udfra det information der er blevet skrevet ud hvad tror du datasættet indeholder?
Tænk over følgende
- Hvor mange kolonner er der?
- Hvad er titlen på hver kolonne?
- Hvor mange rækker er der?
### Opgave 4: Plot datasæt
Nu hvor vi har fået hentet datasættet vil vi gerne undersøge det nemmere ved at
plotte det.
Færdiggør koden nedenfor
```{python}
#| exercise: true
#| eval: false
fig, ax = plt.subplots()
ax.plot(df['time'], ..., label='[A]') # Erstat ... med din kode
... # Erstat ... med din kode som plotter tid mod concentration_B
ax.set_ylabel('Concentration') # Sætter navn på y-aksen
ax.set_xlabel('Time') # Sætter navn på x-aksen
ax.legend()# Viser hvad 'label' hver plottet kurve har.
```
```{python}
#| solution: true
fig, ax = plt.subplots()
ax.plot(df['time'], df['concentration_A'], label='[A]')
ax.plot(df['time'], df['concentration_B'], label='[B]')
ax.set_ylabel('Concentration') # Sætter navn på y-aksen
ax.set_xlabel('Time') # Sætter navn på x-aksen
ax.legend() # Viser hvad 'label' hver plottet kurve har.
```
Hvad tror du datasættet viser?
### Opgave 5: Beregninger med datasæt
Som sagt kan vi også lave beregninger med et datasæt. Udregn
$$
[T] = [A] + [B]
$$
Og færdiggør plottet i cellen nedenfor
```{python}
#| exercise: true
#| eval: false
concentration_T = ... # Erstat ... med din kode
fig, ax = plt.subplots()
ax.plot(..., ..., label='[T]', color='C2') # Erstat begge ... med din kode.
# Fra før
ax.plot(df['time'], df['concentration_A'], label='[A]')
ax.plot(df['time'], df['concentration_B'], label='[B]')
ax.set_ylabel('Concentration') # Sætter navn på y-aksen
ax.set_xlabel('Time') # Sætter navn på x-aksen
ax.legend() # Viser hvad 'label' hver plottet kurve har.
```
```{python}
#| solution: true
concentration_T = df['concentration_A'] + df['concentration_B']
fig, ax = plt.subplots()
ax.plot(df['time'], concentration_T, label='[T]', color='C2')
# Fra før
ax.plot(df['time'], df['concentration_A'], label='[A]')
ax.plot(df['time'], df['concentration_B'], label='[B]')
ax.set_ylabel('Concentration') # Sætter navn på y-aksen
ax.set_xlabel('Time') # Sætter navn på x-aksen
ax.legend() # Viser hvad 'label' hver plottet kurve har.
```
Hvorfor tror du det ser ud som det gør?