Code
try:
import fysisk_biokemi
print("Already installed")
except ImportError:
%pip install -q "fysisk_biokemi[colab] @ git+https://github.com/au-mbg/fysisk-biokemi.git"Forrige gang så vi på funktioner, vi vil fortsætte med det denne gang. Husk at en funktion er en opskrift der beskriver hvordan en beregning skal foretages som kan genbruges mange gange.
En funktion defineres med def hvorefter dens navn bliver sat og i paranteserne sættes dens parametre (også kaldet argumenter).
Funktionen bliver først udført når den bliver kaldt, i dette tilfælde f.eks.
Hvor vi så har outputtet af funktionen i variablen mit_resultat.
Tablene nedenfor viser radius af forskellige celler typer
| Celle type | Radius |
|---|---|
| Mycoplasma | ~0,2–0,3 µm |
| Typisk bakterie | ~1–2 µm |
| Gærcelle (S. cerevisiae) | ~5–7 µm |
| Røde blodlegemer (menneske) | ~7–8 µm |
| Lymfocyt (hvidt blodlegeme) | ~8–10 µm |
| Typisk dyrecelle | ~10–20 µm |
| Typisk plantecelle | ~20–100 µm |
| Menneskelig ægcelle (oocyt) | ~120–140 µm |
| Neuron (cellekrop) | ~10–100 µm |
Vi har tidligere beregnet volumen af cellerne ved at antage at de er kugleformede, hvorved at volumen er givet ved
\[ V(r) = \frac{4}{3}\pi r^3 \]
Skriv en funktion den tager radius r som argument og beregner volumen - husk at du kan bruge np.pi istedet for at skrive \(\pi\) ind selv!
Kald funktionen med nogle af de forskellige radius fra tablen
Vi har tidligere set nogle af forskellige datatyper i Python
intfloatstrMen der er mange andre typer der kan være hjælpesomme i forskellige sammenhæng. En sådan type er numpy arrays, et eksempel på et numpy array er vist nedenfor
Noget af det smarte med arrays er at vi kan lave operationer på alt indeholdet på engang
Lav et array der indeholder de angivne celle radii og brug din volumen funktion til at beregne volumen af alle celler på engang
Der er mange andre operationer vi kan lave på arrays, som f.eks. at finde det største eller mindste tal
Vi kan finde celle-typen med den største volumen som
Den tilsvarende funktioner for at finde det mindste tal hedder np.min.
Cellen nedenfor laver et array med 1000 tal
Brug np.max til at finde det største tal i array_med_tal
Nogle gange er vi intereserede i specifikke indgange i et array, så vi skal også kunne trække data ud af et array. Dette er “indexing” (indeksering), f.eks. hvis vi har et array
Kan vi bruge trække det fjerde tal ud sådan
I Python starter man med at tælle fra 0, så det fjerde tal findes på plads 3.
Brug indexing til at regne summen af
I molekylær biologi arbejder vi ofte med spektra, hvor akserne f.eks. er
Wavelength(nm) AdsorptionCoefficient
0 300 20487.000
1 301 20547.000
2 302 20642.000
3 303 20767.000
4 304 20899.000
.. ... ...
496 796 26.665
497 797 15.556
498 798 29.164
499 799 21.473
500 800 18.396
[501 rows x 2 columns]Text(0, 0.5, 'Adsorption Coefficient')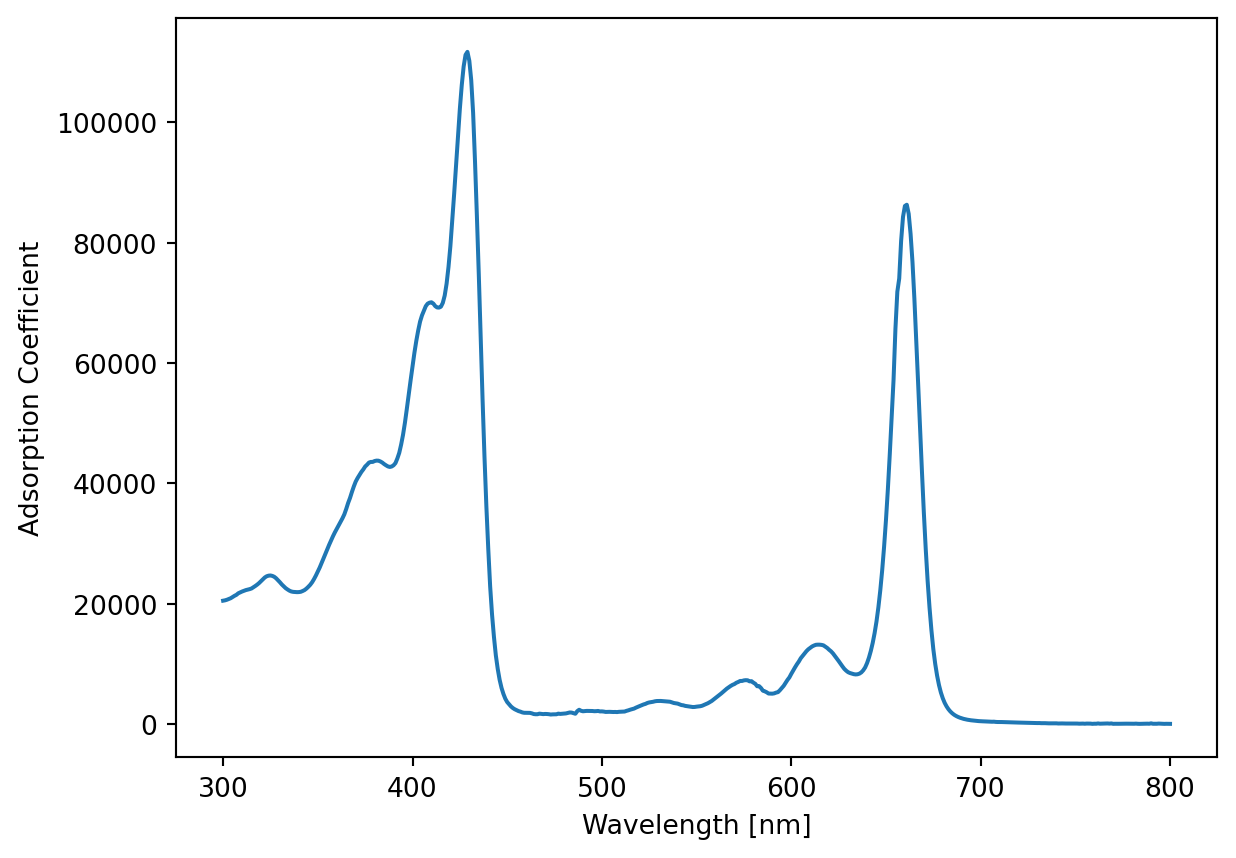
Vi vil gerne bruge NumPy til at finde hvad den maksimale adsorptions koefficient er, og ved hvilken bølgelængde det forekommer.
Start med at finde den maksimale adsorptions koefficient ved brug af np.max
Vi kan bruge en anden funktion fra NumPy til at finde den bølgelængde hvor adsorptions koefficienter har sit maxima
np.argmax finder indekset af det argument som indeholder max værdien.
Brug nu index_max_ads til at trække den pågældende bølgelængde ud af bølgelængder
---
title: Funktioner og arrays
jupyter: python3
categories: ["functions", "arrays"]
format-links:
- text: "Open in Google Colab"
href: "https://colab.research.google.com/github/au-mbg/fysisk-biokemi/blob/built-notebooks/built_notebooks/student/notebook_2.ipynb"
icon: box-arrow-up-right
---
``` {python}
#| eval: false
#| code-fold: true
try:
import fysisk_biokemi
print("Already installed")
except ImportError:
%pip install -q "fysisk_biokemi[colab] @ git+https://github.com/au-mbg/fysisk-biokemi.git"
```
```{python}
import numpy as np
```
## Funktioner
Forrige gang så vi på funktioner, vi vil fortsætte med det denne gang. Husk at en
funktion er en opskrift der beskriver hvordan en beregning skal foretages som
kan genbruges mange gange.
En funktion defineres med `def` hvorefter dens navn bliver sat og i paranteserne
sættes dens parametre (også kaldet argumenter).
```{python}
def min_funktion(a, b):
resultat = a * b
return resultat
```
Funktionen bliver først udført når den bliver kaldt, i dette tilfælde f.eks.
```{python}
mit_resultat = min_funktion(6, 7)
print(mit_resultat)
```
Hvor vi så har outputtet af funktionen i variablen `mit_resultat`.
### Opgave 1
Tablene nedenfor viser radius af forskellige celler typer
| Celle type | Radius |
|---------|:-----|
|Mycoplasma | ~0,2–0,3 µm |
|Typisk bakterie | ~1–2 µm |
|Gærcelle (S. cerevisiae) | ~5–7 µm |
|Røde blodlegemer (menneske) | ~7–8 µm |
|Lymfocyt (hvidt blodlegeme) | ~8–10 µm |
|Typisk dyrecelle |~10–20 µm |
|Typisk plantecelle | ~20–100 µm |
|Menneskelig ægcelle (oocyt) | ~120–140 µm |
|Neuron (cellekrop) | ~10–100 µm |
: Celle størrelser
Vi har tidligere beregnet volumen af cellerne ved at antage at de er kugleformede, hvorved
at volumen er givet ved
$$
V(r) = \frac{4}{3}\pi r^3
$$
Skriv en funktion den tager radius `r` som argument og beregner volumen - husk at
du kan bruge `np.pi` istedet for at skrive $\pi$ ind selv!
```{python}
#| exercise: true
#| eval: false
def ... # Erstat med din kode
... # Erstat med din kode
```
```{python}
#| solution: true
def volumen(r):
return 4/3 * np.pi * r**3
```
Kald funktionen med nogle af de forskellige radius fra tablen
```{python}
#| exercise: true
#| eval: false
mycoplasma_volume = ... # Erstat med din kode
print(mycoplasma_volume)
# Gør det samme for nogle af de andre celle typer
...
```
```{python}
#| solution: true
mycoplasma_volume = volumen(0.3)
print(mycoplasma_volume)
```
## Arrays
Vi har tidligere set nogle af forskellige datatyper i Python
- Integers (Heltal) `int`
- Floats (Kommatal) `float`
- Strings (Tekst) `str`
Men der er mange andre typer der kan være hjælpesomme i forskellige sammenhæng.
En sådan type er numpy arrays, et eksempel på et numpy array er vist nedenfor
```{python}
A = np.array([1.0, 2.5, 3.5])
```
Noget af det smarte med arrays er at vi kan lave operationer på alt indeholdet på engang
```{python}
B = 2 * A
print(B)
```
### Opgave 2
Lav et array der indeholder de angivne celle radii og brug din `volumen` funktion til
at beregne volumen af alle celler på engang
```{python}
#| exercise: true
#| eval: false
radii = np.array([...]) # Erstat ... med din kode
```
```{python}
#| exercise: true
#| eval: false
celle_volumner = ... # Erstat ... med din kode.
print(celle_volumner)
```
```{python}
#| solution: true
radii = np.array([0.2, 1, 6, 7.5, 9, 15, 60, 130, 50]) # Erstat ... med din kode
```
```{python}
#| solution: true
celle_volumner = volumen(radii)
print(celle_volumner)
```
## Array operationer
Der er mange andre operationer vi kan lave på arrays, som f.eks. at finde det
største eller mindste tal
Vi kan finde celle-typen med den største volumen som
```{python}
np.max(celle_volumner)
```
Den tilsvarende funktioner for at finde det mindste tal hedder `np.min`.
### Opgave 3
Cellen nedenfor laver et array med 1000 tal
```{python}
array_med_tal = np.random.rand(1000) * np.exp(3.737669)
```
Brug `np.max` til at finde det største tal i `array_med_tal`
```{python}
#| exercise: true
#| eval: false
største_tal = ... # Erstat ... med din kode
print(største_tal)
```
```{python}
#| solution: true
største_tal = np.max(array_med_tal)
print(største_tal)
```
## Array indeksering
Nogle gange er vi intereserede i specifikke indgange i et array, så vi skal
også kunne trække data ud af et array. Dette er "indexing" (indeksering), f.eks.
hvis vi har et array
```{python}
mit_array = np.array([0, 2, 4, 6, 8, 10, 12, 14, 16, 18, 20])
```
Kan vi bruge trække det fjerde tal ud sådan
```{python}
fjerde_tal = mit_array[3]
```
::: {.callout-important}
## Vigtigt
I Python starter man med at tælle fra 0, så det fjerde tal findes på plads 3.
:::
### Opgave 4
Brug indexing til at regne summen af
- Det sidste tal (Plads 10)
- Det andet tal (Plads 1)
- Det fjerde tal (Plads ??)
- Det ottende tal (Plads ??)
```{python}
#| solution: true
sum_af_tal = mit_array[10] + mit_array[1] + mit_array[3] + mit_array[7]
print(sum_af_tal)
```
```{python}
#| exercise: true
#| eval: false
sum_af_tal = ... # Erstat med din kode
print(sum_af_tal)
```
### Opgave 5
I molekylær biologi arbejder vi ofte med spektra, hvor akserne f.eks. er
- $x$: Bølgelængde målt i nm
- $y$: Absorptions koefficient
```{python}
from fysisk_biokemi import load_dataset
df = load_dataset("chlorophyll")
print(df)
bølgelængder = df['Wavelength(nm)'] # Et array med 501 indgange
adsorption = df['AdsorptionCoefficient'] # Et array med 501 indgange
```
```{python}
import matplotlib.pyplot as plt
fig, ax = plt.subplots()
ax.plot(bølgelængder, adsorption)
ax.set_xlabel('Wavelength [nm]')
ax.set_ylabel('Adsorption Coefficient')
```
Vi vil gerne bruge NumPy til at finde hvad den maksimale adsorptions koefficient er,
og ved hvilken bølgelængde det forekommer.
Start med at finde den maksimale adsorptions koefficient ved brug af `np.max`
```{python}
#| solution: true
max_adsorption = np.max(adsorption)
print(max_adsorption)
```
```{python}
#| exercise: true
#| eval: false
max_adsorption = ... # Erstat ... med din kode
print(max_adsorption)
```
Vi kan bruge en anden funktion fra NumPy til at finde den bølgelængde hvor
adsorptions koefficienter har sit maxima
```{python}
index_max_ads = np.argmax(adsorption)
```
::: {.callout-note}
`np.argmax` finder indekset af det **arg**ument som indeholder **max** værdien.
:::
Brug nu `index_max_ads` til at trække den pågældende bølgelængde ud af `bølgelængder`
```{python}
#| exercise: true
#| eval: false
bølgelængde_max_ads = ... # Din kode her
```
```{python}
#| solution: true
bølgelængde_max_ads = bølgelængder[index_max_ads]
```
```{python}
#| exercise: true
#| eval: false
import matplotlib.pyplot as plt
fig, ax = plt.subplots()
ax.plot(bølgelængder, adsorption)
ax.axvline(bølgelængde_max_ads, color='red')
ax.set_xlabel('Wavelength [nm]')
ax.set_ylabel('Adsorption Coefficient')
```
```{python}
#| solution: true
import matplotlib.pyplot as plt
fig, ax = plt.subplots()
ax.plot(bølgelængder, adsorption)
ax.axvline(bølgelængde_max_ads, color='red')
ax.set_xlabel('Wavelength [nm]')
ax.set_ylabel('Adsorption Coefficient')
```